土壤病毒是土壤生物多样性的重要组成部分,数量繁多,在调控微生物多样性、群落组成和养分循环等方面具有重要的生态功能。然而,由于土壤自身的复杂性及其动态变化特征,病毒在土壤生态系统中发挥的功能及其作用机理,一直是该研究领域的薄弱环节,也是当前土壤生物学、微生态学和环境科学等领域的研究热点和前沿。下面一起来了解下土壤病毒研究常用方法有哪些。
大多数土壤病毒研究的第一步工作是需要将吸附在土壤颗粒上的病毒粒子进行洗脱浸提,而10%的柠檬酸钾溶液、10%的牛肉膏、250 mmol/L的甘氨酸溶液和10 mmol/L的焦磷酸钠溶液为最常用的4种土壤病毒洗脱浸提液,其中对于农田土壤病毒洗脱提取,10%的柠檬酸钾溶液效果相对最佳。
通过比较了3种物理分散方法(振荡、涡旋和超声处理)和10种土壤病毒洗脱浸提液,最终确定了从(亚)热带红壤中提取病毒的优化方法。此外,为得到高浓度的土壤病毒提取液,还需要对其进行土壤病毒纯化及富集。目前最常用的土壤病毒纯化及富集方法是聚乙二醇(polyethylene glycol,PEG)沉淀法、CsCl平衡梯度等密度离心法及切向流过滤法(tangential-flow filtration,TFF)。其中,对于土壤病毒粒子的分离、纯化及富集主要采用切向流过滤法,其原理是利用蠕动泵将土壤病毒洗脱液匀速流经特定孔径的过滤膜,从而实现土壤病毒提取液的纯化及浓缩。
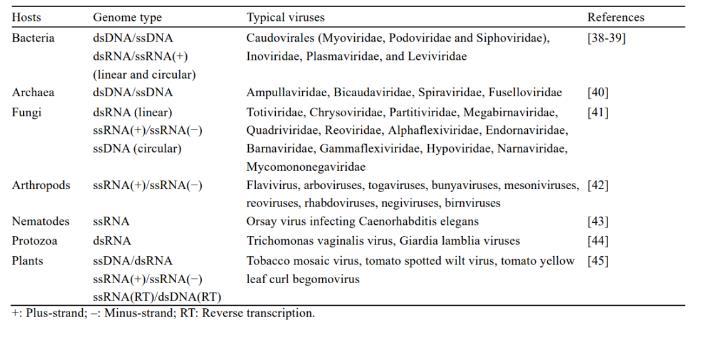
表1土壤典型病毒
在获取纯化及富集后的土壤病毒粒子,可依靠扫描电子显微镜(scanning electron microscope,SEM)、透射电子显微镜(transmission electron microscope,TEM)和流式细胞仪(flow cytometer,FC)等工具,对其进行计数及形态学、生物学特性分析,此外,当前宏病毒组技术也被广泛应用于土壤病毒研究,属于研究热点之一。值得注意的是,由于病毒基因组不同于原核微生物基因组,没有类似于原核生物16S rRNA基因普遍保守的系统发育标记,因此难以利用分子生物技术对土壤病毒多样性和系统发育进行综合分析。
当前唯有在特定病毒类群中,才可利用某些高度保守的标记基因评估某个类群中的系统发育关系(如g23),但携带类似保守基因的病毒只占自然环境中所有病毒种群的极小比例,不能够完整地反映环境中病毒多样性特征。随着高通量测序和宏基因组学技术的发展,人们无须通过宿主培养及单基因标记,仅通过对整个环境中的病毒基因进行比较分析就可以研究病毒多样性。宏病毒组主要有两种分析方法,一是直接收集土壤中的病毒,提取DNA并测序分析。
然而,实验收集的过程会造成一定程度的病毒损失,且由于土壤病毒基因组含量少,需要通过全基因组扩增病毒基因组以达到可测序浓度,会出现偏好性扩增,这些因素都影响着宏基因组序列信息对土壤病毒多样性的真实反映;二是从细菌宏基因组中挖掘病毒序列信息。然而,由于细菌基因组的干扰以及土壤病毒基因库的不完整性,导致土壤病毒的大量序列无法有效注释。由于这些技术方面的限制,目前通过宏病毒组研究土壤病毒多样性的报道较少。
此外,也有研究利用病毒指纹图谱技术来探究土壤病毒多样性,如利用RAPD-PCR(random PCR amplification of polymorphic DNA)技术发现南极不同地点土壤病毒群落结构存在显著差异,且病毒群落结构随环境变化而异。
相关新闻推荐
1、新型广谱阴沟肠杆菌噬菌体ZX14的分离鉴定、生长曲线、体体外杀菌活性实验(三)
