2结果与讨论
2.1 SCRS数据处理和数据质量评估
本文应用HOOKE intP软件对实验组和验证组同步培养的SCRS数据进行批处理。在实验组(大肠杆菌)中,1、2、…、14 h每个培养时间点各采集100个SCRS数据,依据图1中OD600生长曲线分别将1和2 h、3和4 h、6和14 h采集的大肠杆菌SCRS数据对应到lag phase、log phase和stationary phase三个生长时期标签,每个生长时期200个数据。用堆叠图(stacked lines by Y offsets)显示三个生长时期SCRS数据预处理效果,如图2(a)所示,分别以实线和阴影部分显示三个生长时期200个SCRS数据平均值和方差,横坐标为拉曼位移(cm-1),由于微生物生长过程中的异质性较为稳定,表现出三个生长时期光谱具有较低的方差。对三组大肠杆菌的SCRS数据做探索性数据分析(EDA),分别用图2(b)密度图和图2(c)带抖动点的箱线图观测三组数据信噪比(SNR)分布情况,其中lag phase光谱信噪比均值和方差为4.97±1.54,log phase光谱信噪比4.74±1.17,stationary phase光谱信噪比4.84±1.21,三个生长时期SCRS数据特征呈现较为稳定的均匀分布,保证了预期检测结果不受SNR影响。
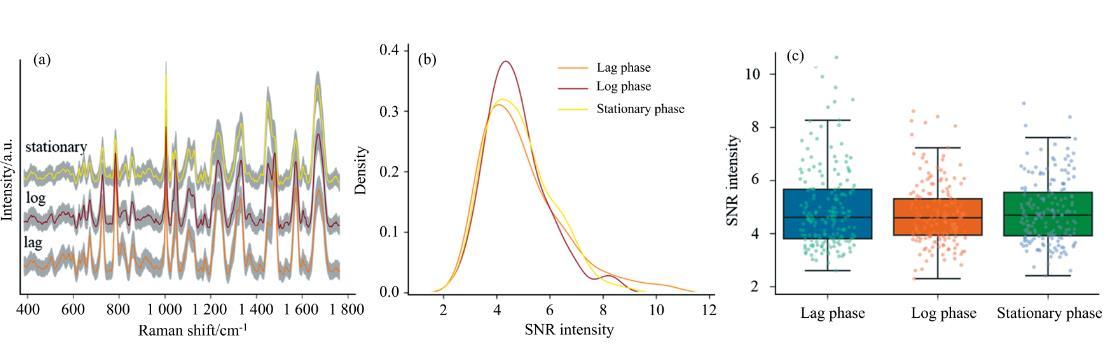
图2大肠杆菌不同生长时期SCRS数据预处理效果
(a):拉曼光谱堆叠图;(b):SNR的密度直方图;(c):带有抖动点的箱线图
2.2基于谱聚类与SCRS的细胞生长检测
基于谱聚类与SCRS的细胞生长检测结果建立在1.3方法的基础上,在t-SNE方法中,嵌入空间维度(n_components)选择为2维,谱聚类的相似度计算方法(affinity)选用最近邻算法,聚类评估中聚类簇数(n_clusters)最大值为9簇。
2.2.1实验组聚类和评估
对实验组600个(6个培养时间点各采集100个SCRS数据)大肠杆菌SCRS数据聚类分析,首先,将高维的SCRS数据应用t-SNE投影到二维平面,见图3(a)中,用不同形状、颜色散点标记同步培养的1、2、3、4、6和14 h等6个生长时期标签的大肠杆菌群体细胞;其次,基于图3(a)的散点分布结果,应用谱聚类对平面上SCRS数据进行聚类分析,见图3(c)中,(c)左下折线图为应用轮廓系数(S_C)和CH index(C—H)对谱聚类在大肠杆菌SCRS数据集上划分的簇数和聚类质量的评估得分折线图,发现当聚为3簇时达到最佳聚类效果,沿着TSNE1和TSNE2坐标分布显示了3个清晰可分离的簇,聚类中心(红色圆点)到簇内和其他聚类中心平均距离(从左到右):(13.86,40.16),(14.16,56.31),(13.98,58.52);最后,应用三次样条插值拟合统计SCRS数据簇标签和OD600生长时期标签交集,图3(b)中有效识别60个异质SCRS数据,占总SCRS数量的9%。
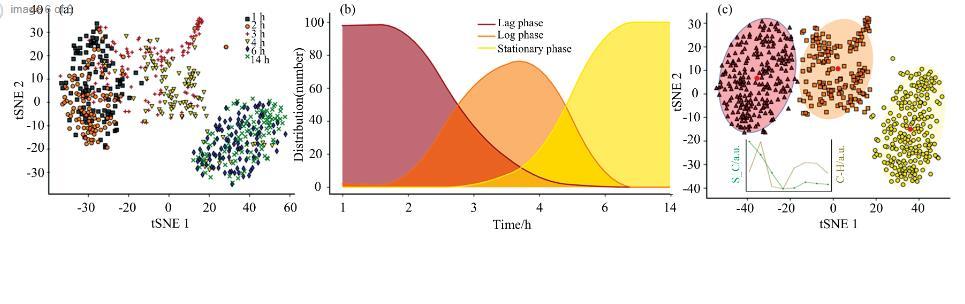
图3应用谱聚类检测大肠杆菌细胞生长时期结果
(a):实验组SCRS的散点分布;(b):三次样条插值拟合效果;(c):SCRS的聚类和评估
2.2.2验证组聚类和评估
用验证组的300个(6个培养时间点各采集50个SCRS数据)枯草芽孢杆菌SCRS数据验证方法适用性,应用与实验组相同的预处理方法,对三组枯草芽孢杆菌的SCRS数据做EDA分析,lag phase、log phase和stationary phase光谱信噪比均值和方差分别为:5.35±0.67、4.85±0.77、5.9±1.01,满足数据质量评估。图4(a)为同步培养下1、2、3、5、8和14 h等6个时期枯草芽孢杆菌SCRS数据经t-SNE压缩后的平面分布;图4(c)轮廓系数(S_C)和CH index(C—H)聚类评估得分显示,不同生长时期的芽孢杆菌同样聚为3簇时达到最佳聚类效果,各聚类中心到簇内和其他聚类中心平均距离(从左到右):(11.82,34.23),(10.23,51.47),(10.01,48.09);图4(b)同样应用三次样条插值拟合统计SCRS数据簇标签和OD600生长时期标签交集,检测出13个不同生长时期异质SCRS数据,占总SCRS数量的4.3%。
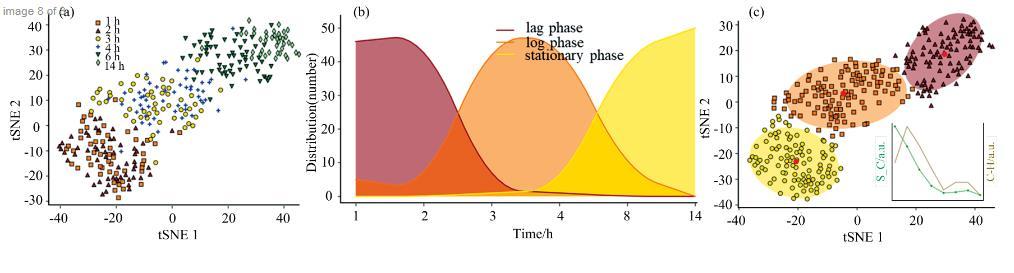
图4应用谱聚类检测枯草芽孢杆菌细胞生长时期结果
(a):验证组SCRS的散点分布;(b):三次样条插值拟合效果;(c):SCRS的聚类和评估
实验和验证结果表明,基于谱聚类与SCRS的细胞生长分析方法只需要借助同步培养的群体细胞OD600生长曲线和给定相似度计算方法就可以直接根据SCRS数据特征进行建模,能有效检测微生物群体中不同生长时期共存的单细胞信息,真正意义上实现从单细胞尺度精准检测细胞生长时期。
3结论
单细胞拉曼光谱技术以快速、灵敏和无标记的优势可以实时监测单细胞的生长代谢变化,以监督学习为代表的模式识别技术往往需要精准的监督标签,然而由于细胞异质性,同步培养的群体细胞OD600生长曲线无法作为每个单细胞生长时期标签。本文将SCRS技术和无监督聚类技术相结合,为单细胞微生物生长检测研究提供新的检测方法,基于谱聚类无需标记就可以直接根据SCRS数据特征进行建模,并能够对任意形状的高维SCRS数据聚类且快速收敛的优势,对发酵工程菌和发酵益生菌细胞滞后期、对数期和稳定期的精准识别,实现了真正意义上从单细胞水平上检测细胞生长,为发酵工程提供更加精准、实时的调控指导,具有重要的工程应用价值。
相关新闻推荐
1、微生物生长曲线分析仪揭示假单胞菌对尼古丁降解过程的未解途径
3、志贺氏菌Vi样噬菌体SboM-AG3与沙门氏菌噬菌体ViI的高度基因组同源性(二)
