单细胞微生物生长时期可分为滞后期(lag phase)、对数期(log phase)、稳定期(stationary phase)和衰亡期(apoptosis phase)4个时期,在不同的生长时期表现出不同的代谢和生产能力,由于细胞的异质性,导致了微生物菌落中不同生长时期的细胞共存,传统群体水平上的细胞生长代谢研究,得到的是系统平均值,掩盖了每个细胞的独特性。在单细胞水平上观测细胞生长是非常重要的,在发酵工程中,单细胞的生理状态是决定发酵产品产量的唯一因素,发酵环境随着底物消耗和产物合成不断变化,导致不同生长时期的发酵细胞代谢不同,产量也不同。随着发酵原料成本的增加和环保意识的增强,对发酵过程的精准控制要求越来越高,从单细胞水平上检测发酵细胞生长时期,并进行特定干预措施改造发酵环境,可以使发酵细胞处于最佳的生长和生产状态。准确检测单细胞所处生长时期,可为发酵工程获得最佳产量提供更加精准、实时的调控指导。
单细胞拉曼光谱(single-cell Raman spectroscopy,SCRS)是细胞的指纹图谱,蕴含着细胞在特定生长状态下丰富的表型信息,SCRS技术以快速、灵敏和无标记的优势可以实时监测单细胞的生长代谢变化。以监督学习为代表的模式识别技术通过对SCRS数据学习并生成经验模型,可以指导细胞生长时期鉴定,已有研究人员应用随机森林算法实现了群体水平上的细胞生长检测,相较于监督学习,无监督学习只需要定义相似度计算方法就可以直接根据SCRS数据特征结构进行建模,就能从单细胞尺度上实时监测细胞生长代谢变化。
本文提出基于谱聚类与SCRS的细胞生长分析方法,首先,采集同步培养下不同生长时间的微生物SCRS数据,对应培养时间的OD600生长曲线作为微生物群体水平生长时期标签;其次,应用t分布随机邻居嵌入(t-distributed stochastic neighbor embedding,t-SNE)对群体细胞SCRS数据进行可视化分析,指导谱聚类对高维SCRS数据聚类分析,并应用轮廓系数和CH系数(calinski-harabasz index,CH index)评估最佳聚类簇,赋予每个SCRS数据簇标签;最后,应用三次样条插值拟合统计SCRS数据簇标签和生长时期标签交集,精准识别群体中共存的生长时期异质数据,实现对单细胞微生物生长时期精准鉴定。
1实验部分
1.1 SCRS数据采集
1.1.1分光光度计检测和SCRS检测条件
在细胞培养实验中,提取不同培养时间点各3 mL菌液,应用紫外分光光度计检测,检测条件为OD600,记录细菌生长状态,图1分别为实验组(大肠杆菌)和验证组(枯草芽孢杆菌)各3次重复测量同步培养的OD600生长曲线,将其作为群体水平生长时期标签。同时在各培养时间点提取1μL菌液,应用HOOKE P300共聚焦拉曼光谱仪进行SCRS采集(由于微生物SCRS在600~1 800 cm-1波段具有明显的光谱模式,往往作为其表型指纹区域,故光谱仪主要参数设置为,激发波长(excitation wavelength):532 nm,光栅(Grating):1 200 g·mm-1,激发功率(laser power):3 mW,积分时间(integration time):8 s。SCRS检测可以获取单个细胞生长过程的实时变化信息,提供了用于生物分析的化学组成和结构信息的指纹图谱,蕴含着细胞在特定生长状态下丰富的表型信息,检测SCRS特征峰强变化是细胞生长定性、定量检测的主要依据,可以通过检测细胞代谢活跃的核酸(I785.5、I1 047.5、I1 097.2等)、蛋白(I624.3、I831.2、I1 034等)、脂质(I878、I1 075)等相关特征峰强度变化,实时检测单细胞的生长代谢状态。SCRS是进行单细胞活体生长检测的实用工具,本文将SCRS技术和无监督聚类技术相结合,为单细胞微生物生长检测研究提供新的检测方法。
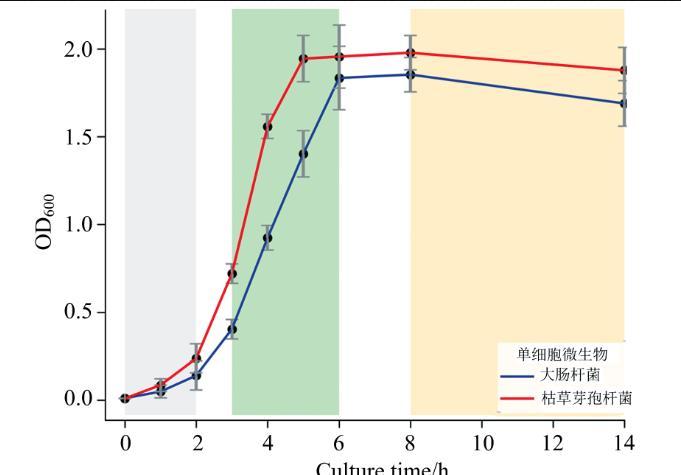
图1单细胞微生物同步培养与OD600生长曲线
1.1.2微生物样品选择与同步培养
将常用的发酵工程菌-大肠杆菌进行同步培养实验,以获取不同生长时期的单细胞样品,作为模式生物突出的代表,大肠杆菌具有繁殖迅速,培养代谢易于控制的优势,是目前生命科学研究最为公认的微生物材料。准确检测发酵过程中工程菌生长状态,是获取最佳发酵产量的前提。为了验证本文方法的适用性,同时选用了一种常用发酵益生菌-枯草芽孢杆菌作为细胞生长研究验证组样品。
大肠杆菌的培养基为LB(luria-bertani),而枯草芽孢杆菌的培养基为牛肉膏蛋白胨,分别在其对应的培养基进行同步培养,记录细菌的培养时间。根据图1不同培养时间OD600生长曲线,确定培养2 h为滞后期,该时期菌体增大,代谢活跃,合成并积累充足的酶和代谢产物;3~4 h为对数期(验证组为3~5 h),细菌在该时期生长迅速,呈现指数生长趋势,增代时间最少;培养6 h(验证组为8 h)至14 h,即进入稳定期,随着培养时间延长,未发现明显的凋亡期界限,但这并不影响生长曲线的走向以及对单细胞生长时期的识别,本文仅涉及细胞前三个时期检测。应用SCRS检测条件分别从实验组和验证组提取的菌液采集SCRS数据,实验组6个培养时间点共采集600个(6时间点×100个/时间点)SCRS数据,验证组6个培养时间点共采集300个(6时间点×50个/时间点)SCRS数据。
1.2 SCRS数据预处理
SCRS数据预处理是准确鉴定细胞生长时期的前提,通过拉曼光谱仪采集的SCRS数据包含大量的干扰信息,如光谱仪噪声、荧光背景等,干扰信息使得检测模型的识别性能降低,在数据分析之前,需要对SCRS数据进行预处理。本文应用HOOKE intP拉曼光谱分析软件对SCRS数据预处理,包括:应用基于Savitzky-Golay卷积平滑对SCRS数据进行滤波处理,窗口宽度为7个光谱像素点,采用三阶多项式拟合;应用基于airPLS(自适应迭代重加权惩罚最小二乘)算法去除拉曼光谱背景信号,Lambda=15,最大迭代次数ItermaxAirPls=12;应用Min-Max对SCRS数据归一化处理。
1.3检测模型
基于谱聚类与SCRS的细胞生长分析方法应用的主要技术包括:(1)应用t-SNE对群体细胞SCRS数据进行可视化分析;(2)应用谱聚类对SCRS数据聚类分析;(3)应用轮廓系数和CH index评估最佳聚类簇。
1.3.1 t-SNE
t-SNE算法是一种适合于高维SCRS数据的非线性降维方法,该方法首先将高维空间中任意两个光谱数据间的欧氏距离转换为相似概率,其次用高维空间数据点与相应低维空间的模拟数据点之间的联合概率替换随机邻域嵌入算法中的条件概率,并在低维空间中使用t分布,有效解决低维空间中数据点拥挤的问题。以二维可视化为例,t-SNE将SCRS指纹特征向具有最大投影信息量的二维平面上投影,以高维空间相同概率分布的TSNE1和TSNE2特征分量均匀分布在平面中,有效解决低维空间中数据点拥挤的问题,用于直观显示不同培养时间单细胞分布效果,指导无监督聚类分析。
1.3.2谱聚类
谱聚类(spectral clustering)是一种基于两点间相似关系的无监督聚类算法,首先对SCRS数据样本高维矩阵进行低维嵌入,然后进行聚类。其本质是将聚类问题转化为图的最优划分问题,相较于其他传统聚类算法,谱聚类能在任意形状的SCRS数据样本空间上聚类且易于收敛到全局最优,并且通过构造稀疏相似性图谱,使其对于高维SCRS数据集表现出更快的计算速度。特别的,相较于监督学习的分类算法,谱聚类无需SCRS数据标签,只需要定义相似度计算方法就可以直接根据SCRS数据特征进行建模,能有效检测微生物群体中不同生长时期共存的单细胞信息。
1.3.3聚类评估
聚类评估是对聚类方法产生结果的质量进行评估,主要任务包括:估计聚类趋势、确定数据集划分簇数以及评估聚类质量,应用轮廓系数和CH index两个维度来评估谱聚类在SCRS数据集划分最佳簇数和聚类质量。
(1)轮廓系数(silhouette coefficient)是聚类效果好坏的一种评价方式,它结合内聚度和分离度两种因素,在SCRS数据上评价谱聚类对聚类结果所产生的影响,式(1)是轮廓系数聚类得分计算原理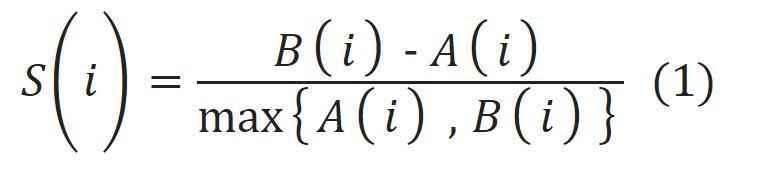
式(1)中,A(i)为SCRS数据样本i到同簇其他样本的平均距离,B(i)为SCRS数据样本i到其他簇的所有样本的平均距离。
(2)CH index也被称为方差比准则,用来评价谱聚类在SCRS数据集上的聚类效果,聚类质量由CH index得分表征,CH index得分通过计算簇间方差和簇内方差计算得到的,式(2)是CH index得分计算原理(2)
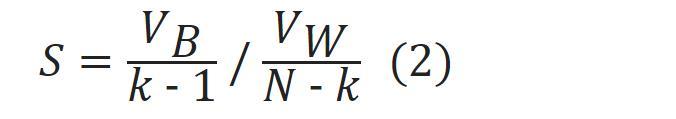
式(2)中,k为谱聚类在SCRS数据集上聚类簇数,N为全部SCRS数据样本个数,VB是簇间方差,VW是簇内方差。
相关新闻推荐
2、类肠膜魏斯氏菌对猪生长性能、粪菌和代谢产物的影响——摘要、材料与方法
