本研究揭示了插入序列(IS)在原核生物基因组中的转座如何导致CRISPR-Cas免疫系统的失活。CRISPR-Cas系统是原核生物用来防御外来遗传元素的主要机制,通过特定的导向RNA(crRNA)指导Cas蛋白识别并切割目标序列。研究者通过筛选原核生物基因组序列,发现了多个自然发生的IS插入事件,这些事件破坏了CRISPR-Cas位点,尤其是IS1和IS10,它们显示出对靶标特异性的显著放宽。本研究构建了一个IS捕获系统,监测在双链DNA断裂诱导后IS插入到cas基因的事件,发现IS转座在DNA修复机制存在的情况下仍然可以发生,并且IS转座也检测到其他宿主防御系统的插入。这些发现强调了IS在抵消CRISPR-Cas活性、增加细菌对外国DNA入侵的易感性方面的潜力,这可能有助于细菌获取有益的外源性移动遗传元素(MGEs),增强环境适应性。研究还探讨了IS在细菌适应性中的角色,包括它们如何通过破坏CRISPR-Cas系统来促进有益MGEs的获取,以及这种机制如何影响细菌的进化。通过异源表达特定类型的CRISPR-Cas系统,并构建含有特定原间隔序列的质粒,研究验证了IS插入对CRISPR-Cas系统的干扰作用。这项研究不仅增进了对CRISPR-Cas系统和IS之间相互作用的理解,还对抗生素抗性传播和细菌进化的研究具有重要意义。
Bioscreen全自动微生物生长曲线分析仪的应用
Bioscreen全自动生长曲线分析仪用于测定不同TIR防御系统在大肠杆菌中表达时对噬菌体感染的反应。通过将单个转化体在37°C下用适当的抗生素在LB培养基中培养过夜,然后以1:100稀释并接种到含有400μL LB肉汤和不同抗生素浓度的Bioscreen 100孔蜂窝板中。在37°C下每20分钟监测一次OD 600,持续25小时,并在Bioscreen C设备(Bioscreen C Labsystems)中连续摇动。通过测量细菌的吸光度(OD600),了解噬菌体感染对细菌生长的影响,以及TIR系统是否有效抑制了噬菌体的增殖。Bioscreen被用于快速筛选和鉴定那些能够逃避TIR防御系统的噬菌体。通过连续监测细菌培养的生长情况,研究人员可以识别出在TIR防御系统存在下仍能生长的噬菌体,这些噬菌体可能是通过突变获得了逃避TIR系统的能力。
实验结果
本研究强调了插入序列(IS)在细菌适应性进化中的关键作用。研究发现,IS元件能够通过破坏CRISPR-Cas免疫系统,增加细菌对外国DNA的易感性,从而促进有益外源基因的获取。这种机制揭示了细菌在遗传防御和外源基因获取之间的进化权衡关系。具体来说,IS元件,尤其是IS1和IS10,通过转座至cas基因,显著放宽了靶标特异性,导致CRISPR-Cas系统的失活。即便在DNA修复机制存在的情况下,IS转座至cas基因的活动仍然持续,并且这种转座活动也影响到了其他宿主防御系统。这一发现不仅拓宽了我们对CRISPR-Cas失活机制的理解,而且对于深入理解细菌遗传多样性、定殖、生存和进化策略提供了新的视角。
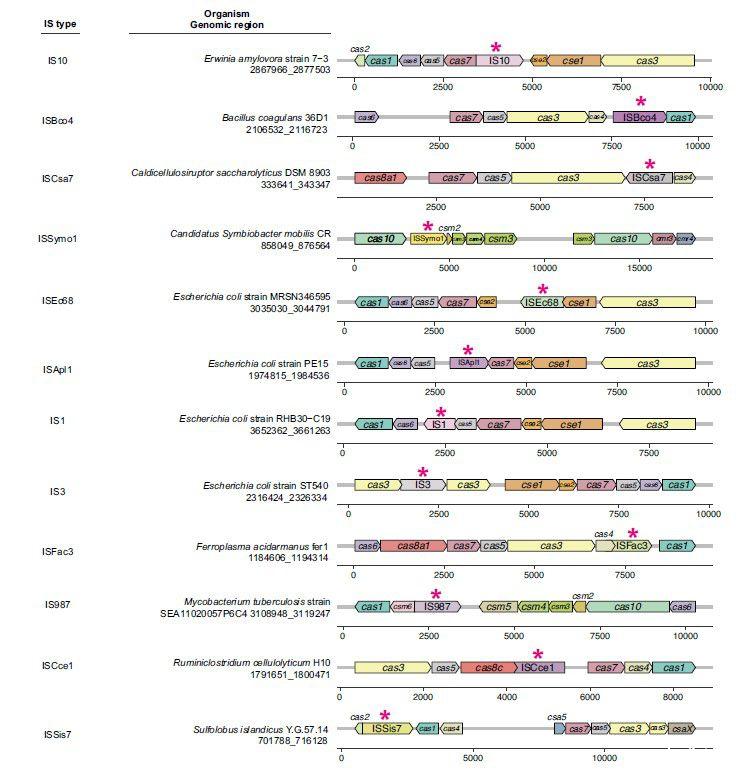
图1、IS元件向CRISPR-Cas基因座的天然转座。显示了在CRISPR-Cas基因座中发现的每个IS元件的代表性示例。箭头代表CRISPR-Cas簇内的IS(用星号标记)和基因,箭头方向表示IS插入或基因转录的方向。物种名称下方的基因组坐标表示每个cas簇的范围,编号线表示每个基因或IS在相应区域内的相对位置。
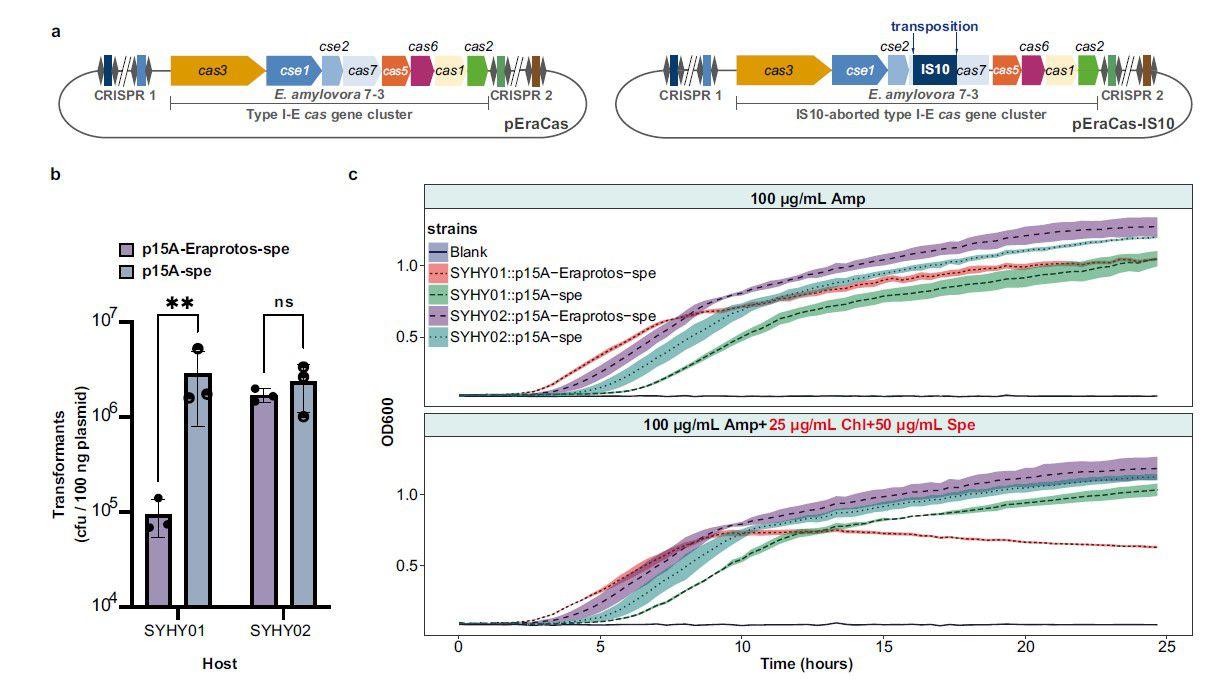
图2、IS失活的CRISPR-Cas系统有助于快速获取和提高含有可识别原始间隔区的抗生素抗性质粒的持久性。a)E.amylovora 7-3的工程型I-E CRISPR-Cas基因座示意图。将完整或IS10中止的CRISPR-Cas基因座分别克隆到质粒pEraCas或pEraCas-IS10中,用于CRISPR-Cas干扰测定。b)E.amylovora 7-3型I-E CRISPR-CAS系统对大肠杆菌DH10B细胞的质粒转化干扰。通过计算每100 ng质粒的集落形成单位(CFU)来确定转化效率。数据显示为来自三个生物学重复的±SD平均值;**P<0.01;ns不显著,使用log10转换数据的双尾未配对t检验;P=0.0018,0.6150(从左到右)。源数据作为源数据文件提供。c)不同抗生素暴露下供试菌株的生长曲线。阴影区域表示3次生物学重复±S.D.的平均值。氨苄青霉素(Amp)用于维持宿主体内的pEraCas或pEraCas-IS10,氯霉素(Chl)和壮观霉素(Spe)用于维持p15A-Eraprotos-spe和p15A-spe。
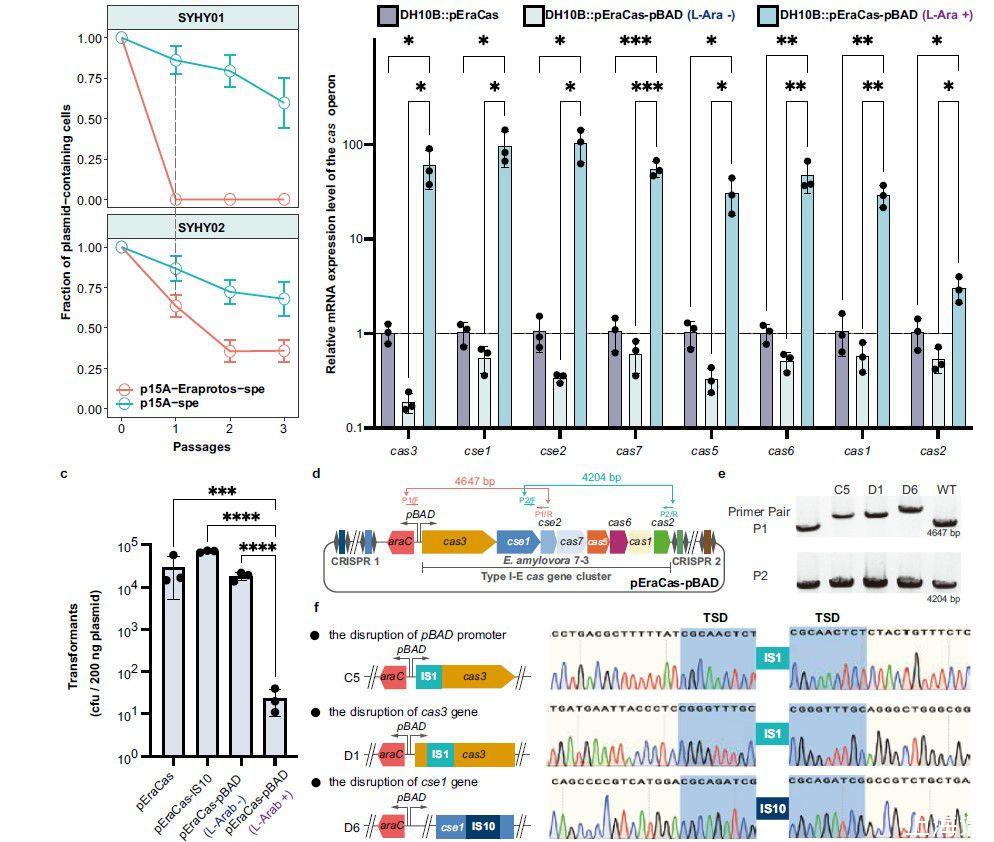
图3、IS介导了在选择压力下获得质粒的好处和遗传denfense之间的适应性权衡。a)在SYHY01和SYHY02中转化和传代期间的质粒稳定性。b)大肠杆菌DH10B中pEraCas和pEraCas-pBAD的cas操纵子内8个基因的相对转录水平。通过RT-qPCR定量基因表达并归一化为内源性16sRNA水平。c)通过计数每200 ng质粒的CFU来确定pEraCas(靶向SYHY03染色体)、pEraCas-IS10(对照)和pEraCas-pBAD(在l-阿拉伯糖存在下靶向SYHY03染色体)向SYHY03的转化效率。d)pEraCas-pBAD携带L-阿拉伯糖诱导型pBAD启动子以驱动cas操纵子表达的示意图。引物对P1和P2旨在扩增pEraCas-pBAD的整个cas操纵子;显示了预期的扩增子大小。e使用引物对P1和P2对IS插入pEraCas-pBAD cas操纵子进行菌落PCR筛选。泳道“WT”,用pEraCas-pBAD模板对照扩增子;较大的PCR产物(泳道C5、D1和D6)提示IS转座事件。f)通过Sanger测序确认IS插入突变体C5、D1和D6的cas操纵子中。
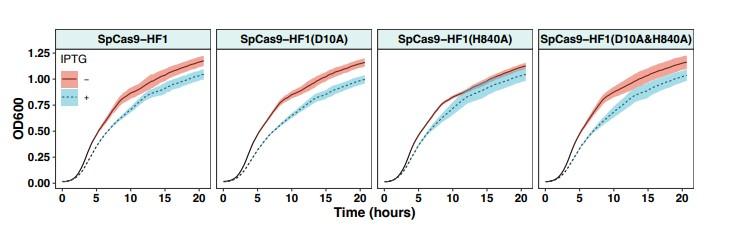
图4、仅诱导Cas蛋白表达不会导致宿主菌株出现明显的生长缺陷。在IPTG诱导和非诱导条件下,编码不同Cas蛋白变体的菌株的生长曲线。
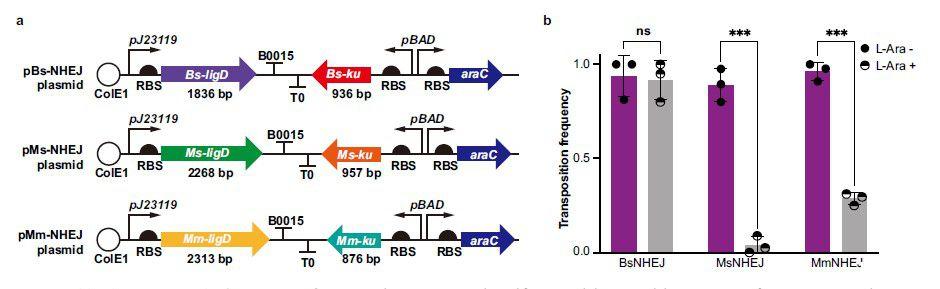
图5、在NHEJ存在下IS转座到cas基因中。a)示意图显示了三个NHEJ系统的组装,其中包括参与NHEJ修复的ku和ligD基因。b)在含有ompF(由SpCas9-HF1/sgRNA介导的DSB靶基因)的MG1655-ΔrecA中测定转座频率,然后进行易错的NHEJ DNA修复。添加l-阿拉伯糖(10 mM)诱导ku表达。每个板最多随机选择45个存活菌落进行PCR验证,并计算每种条件下IS插入SpCas9-HF1编码区的比例。
总结
CRISPR-Cas免疫系统通过抑制移动遗传元件的入侵来保护原核基因组。研究人员筛选了原核基因组序列,并鉴定了插入序列(IS)到cas基因中的多个自然转座,从而灭活CRISPR-Cas防御。使用具有各种IS和诱导型cas核酸酶的大肠杆菌菌株生成了一个IS捕获系统,以监测作为生理宿主应激诱导双链DNA断裂后IS插入cas基因的情况。研究人员鉴定了由不同ISs介导的多个事件,尤其是IS1和IS10,显示出显著的松弛靶标特异性。在DNA修复机制存在的情况下,IS转座到cas中得以维持,并且还检测到转座到其他宿主防御系统。Bioscreen全自动生长曲线分析仪用于测定不同TIR防御系统在大肠杆菌中表达时对噬菌体感染的反应。通过连续监测细菌培养的生长情况,研究人员可以识别出在TIR防御系统存在下仍能生长的噬菌体,这些噬菌体可能是通过突变获得了逃避TIR系统的能力。Bioscreen的使用使得研究人员能够自动化地完成这些测量和分析,大大提高了实验的效率和准确性。本研究结果强调了IS对抗CRISPR活性的潜力,从而增加细菌对外源DNA侵袭的易感性。研究还探讨了IS在细菌适应性中的角色,包括它们如何通过破坏CRISPR-Cas系统来促进有益MGEs的获取,以及这种机制如何影响细菌的进化。通过异源表达特定类型的CRISPR-Cas系统,并构建含有特定原间隔序列的质粒,研究验证了IS插入对CRISPR-Cas系统的干扰作用。这项研究不仅增进了我们对CRISPR-Cas系统和IS之间相互作用的理解,还对抗生素抗性传播和细菌进化的研究具有重要意义。
相关新闻推荐
1、嗜碱盐单胞菌菌株生理生化与生长特性、最优发酵条件——讨论、结论
2、老窖泥筛选分离的6株乳酸利用菌培养、发酵性能、生长范围(一)
